DETECCIÓN DE QTLs PARA DEPOSICIÓN DE GRASA Y CRECIMIENTO EN EL CROMOSOMA 4 EN UN CRUCE F2 DE CERDO IBÉRICO Y LANDRACE
Clop A . (1) , Pérez Enciso M. (2) , Sánchez E. (1), Óvilo C. (3), Coll A. (1), Folch J.M. (1), Sánchez A.. (1), Estany J. (2), Babot D. (2), Noguera J.L. (2)(1) Unitat de Genètica i Millora animals, Fac. de Veterinària, Universitat Autònoma deBarcelona, 08193 Bellaterra. (2)Àrea de Producció Animal, Centre UdL-IRTA. E-25198 Lleida. (3)Area de Mejora Genética animal, CIT-INIA, 28040 Madrid.
INTRODUCCIÓN
La detección de QTLs (Quantitative Trait Loci), regiones del genoma que intervienen en el valor cuantitativo de un determinado carácter, ha sido objeto de un creciente interés en los últimos años a partir de la existencia de marcadores moleculares (microsatélites, AFLPs, etc...) en el genoma de las principales especies domésticas. En el mapa genético porcino existen numerosos microsatélites bien localizados y altamente polimórficos (Rohrer et al., 1996).
Se realizó un cruce experimental tipo F2 de cerdos de la raza Ibérica con cerdas Landrace con el objetivo de detectar marcadores moleculares ligados a caracteres de interés productivo en porcino. Estas dos poblaciones tienen características bien diferenciadas en cuanto a la calidad de la carne (IBMap Consortium, 1998 y Serra et al., 1998). El cruce entre individuos fenotípicamente muy distanciados incrementa la potencia estadística de este tipo de diseño.
En este trabajo presentamos los datos relativos a la detección de QTLs en el cromosoma 4 porcino, con efecto sobre la deposición de grasa y el crecimiento, dos caracteres de importancia para la industria porcina. Paralelamente, se ha realizado con el mismo material animal, un estudio sobre QTLs que afectan a la calidad de la carne en el cromosoma 6 porcino (Óvilo et al., 1999).
MATERIAL Y MÉTODOS
Animales. Tres machos Ibéricos pertenecientes a la línea Guadyerbas fueron cruzados con 31 hembras Landrace homocigotas dominantes en el diagnóstico genético del gen Hal (Sánchez et al., 1993). La F1 está formada por 6 machos y 73 hembras que han generado 577 individuos F2. El manejo de los animales ha sido el habitual en condiciones comerciales.
En este trabajo presentamos los resultados correspondientes al análisis de 247 F2, agrupados en 33 familias de hermanos. El sacrificio se llevó a cabo en 4 lotes a 175.5 ± 0.3 días de edad y un peso de canal promedio de 74.7 ± 0.6 Kg. Los caracteres estudiados fueron el peso de la canal corregido por edad al sacrificio (P.C.), espesor de grasa entre la 3ª y 4ª costilla y a unos 6 cm de la línea media (G2), peso de la grasa dorsal en la mitad izquierda de la canal obtenida en un despiece comercial (P.G.D.), y porcentaje de magro (% magro), estimado a partir de G2 y la profundidad del lomo utilizando la ecuación de regresión propuesta por Gispert y Diestre (1994). Estos tres últimos caracteres se corrigieron por peso de la canal.
ADN. La extracción de ADN genómico se realizó a partir de sangre. Para la población parental se utilizó un protocolo de precipitación salina (Miller et al., 1988). Para la F1 y la F2 se utilizó un kit comercial (Boehringer Mannheim) basado, también, en una precipitación salina.
Genotipados. Las reacciones de amplificación de los microsatélites se realizaron en un equipo automatizado de PCR ABI PRISM 877 Integrated Thermal Cycler de Perkin Elmer. Los productos de PCR con cebadores marcados fluorescentemente se analizaron en un equipo de electroforesis capilar y detección fluorescente (ABI PRISM 310 Genetic Analyzer). En la elección de los microsatélites se tuvo en cuenta la posición de éstos en el genoma y su variabilidad, estimada mediante el índice de informatividad de Ron (Ie) (Ron et al., 1995), que fue estimada a partir de una muestra de la población parental (Clop et al., 1998). Para el cromosoma 4, se seleccionaron 7 microsatélites. La construcción posterior del mapa se realizó utilizando el programa Cri-Map (Green et al., 1990) .
Análisis estadístico. El modelo para el análisis de QTLs incluye el sexo, lote y genotipo del QTL como efectos fijos, así como peso canal como covariable, excepto en el análisis del peso canal que se corrigió por la edad al sacrificio. El análisis se realizó mediante mapeo de intervalos con regresión (Haley et al., 1994), que permite incluir la información de todos los marcadores cuando los genotipos no son informativos. Se asume que existe un QTL con alelos alternativos fijados en cada raza con valores a y –a para los homocigotos de cada raza, y d para los heterocigotos. El modelo que incluye el QTL se compara con el modelo sin QTL cada cM mediante un test F clásico.
RESULTADOS Y DISCUSIÓN
Mapa de ligamiento: El mapa resultante de nuestros datos se asemeja al descrito por otros autores (Rohrer et al., 1996) y cubre 121 cM. La longitud del cromosoma 4 es de 144 cM en las hembras y 127,8 cM en los machos. La informatividad de los microsatélites, calculada sobre los individuos de la F2 (contenido informativo de la F2) coincide con la estimada mediante el índice de Ron (Ie) a partir de una muestra de animales de la población parental (Tabla 1). Por lo tanto puede tomarse como criterio para seleccionar microsatélites según su nivel de informatividad.
Tabla 1
Microsatélite |
Posición Rorher et al. (1996) |
Posición (cM) presente trabajo |
Ie |
Contenido Informativo F2 presente trabajo |
SW2404 |
0 |
0 |
0.57 |
0.510 |
S0301 |
27 |
36 |
0.84 |
0.805 |
S0001 |
42 |
55 |
0.89 |
0.844 |
SW839 |
63 |
65 |
0.96 |
0.992 |
S0214 |
81 |
83 |
0.96 |
0.998 |
SW445 |
106 |
105 |
1.00 |
0.948 |
S0097 |
120 |
121 |
0.86 |
0.773 |
La informatividad fue muy alta en 6 de los 7 microsatélites, siendo SW2404 el de menor contenido informativo.
Análisis de QTLs. El valor aditivo a y el dominante d se calcularon sobre la mejor estima de la posición del QTL obtenida mediante el test F clásico (Tabla 2). El valor mínimo de significación de F en otros trabajos similares a éste, se sitúa alrededor de 8 (Andersson et al, 1994; Walling et al, 1998).
Tabla 2
Carácter |
Posición (cM) |
F máx. |
a |
d |
P.C. |
77 |
5.682 |
-2.334±0.859 |
1.805±0.129 |
G2 |
71 |
11.337 |
2.453±0.569 |
-1.073±0.862 |
P. G.D. |
70 |
15.138 |
0.24±0.046 |
-0.046±6.98 |
% magro |
71 |
14.439 |
-2.344±0.471 |
0.813±0.715 |
El peso canal es el único carácter estudiado que no presenta un valor F máximo significativo. Los restantes factores superan claramente el mínimo significativo.
El alelo ibérico implica disminución de P.C. y % magro, mientras que G2 y P.G.D. se ven incrementadas cuando la variante ibérica se encuentra presente. Esto concuerda con que la raza Ibérica es mucho más grasa que la Landrace. En la Figura 1 se muestran los valores de F para cada cM del cromosoma 4, para los caracteres medidos.
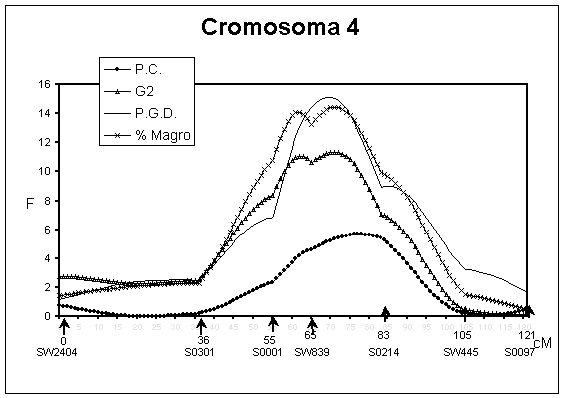
Figura 1
Por la posición cromosómica en que se ha detectado, resulta probable que se trate de un mismo QTL con efecto sobre P.C., G2, P.G.D., y % magro. Asimismo parece razonable pensar que sea el mismo detectado por Andersson et al. (1994) y a su vez, por Walling et al. (1998) en otras poblaciones. No obstante, el valor que adquiere en nuestro estudio el peso canal no es significativo, quizás debido a que los alelos no estén fijados en cada población.
No existen evidencias de un efecto directo sobre alguno de estos caracteres en los genes localizados en este cromosoma. Sin embargo, en la región detectada, hay algunos loci estructurales que podrían tener cierta influencia sobre ellos. Entre éstos se encuentra la subunidad beta 1 de la ATPasa 1 (ATP1B1), los factores de transcripción OTF, SSC9C8, la beta glicosidasa (GBA) y la subunidad beta de la hormona estimulante del tiroides (TSHB).
AGRADECIMIENTOS
Al SIA Dehesón del Encinar que facilitó los parentales Guadyerbas. A Pere Borràs, Eva Ramells y en general a todo el personal de Nova Genètica y del matadero de Copaga por su inestimable cooperación. A Agustí Quintan y Meritxell Arqués por la ayuda en la toma de muestras. A Max Rothschild y al USDA supported US Pig Genome Coordination Project por el generoso envío de cebadores A Chris Haley por los programas de análisis y al resto del equipo IBMAP. Trabajo financiado por la CICYT (proyecto AGF 96-2510). Àlex Clop tiene una predoctoral de formación de investigadores concedida por la Universitat Autònoma de Barcelona.BIBLIOGRAFÍA
-Andersson L. et al.: Science, vol. 263 1771-1774, 1994.
-Archibald A. et al.: Mammalian Genome, 6. 157-175, 1995.
-Clop A. et al.: IX Reunión Nacional de Mejora Genética Animal, 1998. Libro de Resúmenes. ITEA.
-Green P. et al.: Documentation for CRI-MAP, version 2.4, 1990.
-Gispert M., Diestre, A.: Techniporc 17:2 1994.
-Haley C. et al.: Genetics 136, 1195-1207 1994.
-Marklund L. et al.: Animal Genetics, 27. 256-269, 1996.
-Miller S. A. et al.: Nucleic Acid Research, 16, n. 3, 1215.
-Óvilo et al.: ITEA, 1999 (enviado).
-Ron M. et al.: Animal Genetics, 26. 439-441, 1995.
-Rorher G. et al.: Genome Research, 6:371-391, 1996.
-Sánchez et al., ITEA 12: 257-259 1993.
-Serra X. et al., Livestock Production Science 56: 215-232, 1998 .
-The IBMAP Consortium: 6th world Congress on genetics applied to livestock production, 1998.
-Walling G. et al.: Animal Genetics 29. 415-424, 1998.