Evaluación Para Prolificidad En Un Esquema Large White Con Animales Hiperprolíficos
Fernández1, A., Rodríguez1, M.C. y A. Fuentetaja2.
1
Dpto. Mejora Genética y Biotecnología, INIA. Madrid2
COPESE. Conde de Sepulveda 24. Sepulveda, Segovia.
INTRODUCCIÓN
La importación de animales de las líneas hiperprolíficas (HP) Large White y Landrace desarrolladas en diversos países es, actualmente, una práctica común en los esquemas comerciales de mejora para aumentar el tamaño de camada en cerdos (Herment y col. 1994, Noguera y col., 1998). En esquemas abiertos de mejora que emplean animales HP, puede encontrarse individuos con diversa proporción de genes de esta línea. La línea HP y otros posibles orígenes genéticos deben considerarse como grupos genéticos distintos en el modelo de análisis para evitar sesgos en la evaluación genética.
El muestreo de Gibbs permite realizar inferencias Bayesianas acerca de los parámetros de interés en un modelo animal con repetibilidad (Wang y col., 1994). El presente estudio es una aplicación de estas técnicas a datos de prolificidad en un esquema Large White de selección para evaluar las diferencias entre los animales hiperprolíficos importados y la población base.
MATERIAL Y MÉTODOS
Los datos analizados en el presente estudio han sido recogidos, desde 1993 hasta la fecha, en dos granjas incluidas en el esquema Large White de selección de Comercial Pecuaria Segoviana, S.A., que importa continuamente animales hiperprolíficos procedentes de Francia. Un resumen de la información disponible en estos momentos para la evaluación del tamaño de camada se muestra en la Tabla 1.
Tabla 1. Características del conjunto de datos analizados
Nº de animales en genealogía |
3670 |
Animales hiperprolíficos importados |
26 |
Nº de madres con dato |
2436 |
Nº de camadas |
7255 |
Lechones nacidos por camada (dt) |
10.75 (3.43) |
Lechones nacidos vivos por camada (dt) |
9.51 (3.25) |
Los datos han sido analizados con técnicas Bayesianas mediante muestreo de Gibbs. El modelo lineal empleado para el análisis de los datos puede ser representado en notación matricial como:
![]()
![]() ~
~
![]()
![]() ~
~ ![]()
![]() ~
~ ![]()
siendo:
y = vector de observaciones (lechones nacidos vivos por camada), X = matriz de incidencia de los efectos fijos, b = vector de efectos fijos: ordinal de parto (6 niveles:1, 2, 3, 4, 5, 6 o más); tipo de cruce (3 niveles: LW x LW, LD x LW y PT x LW); granja (2 niveles) y año – estación (24 niveles), Z, W = matrices de incidencia de valores genéticos aditivos y efectos permanentes, a, c, e = vectores de valores genéticos aditivos, efectos permanentes y residuales, s 2a, s 2c, s 2e= varianza aditiva, de efecto permanente y residual, A = matriz de parentesco, Q = matriz de elementos qij, proporción de genes del animal i procedentes del grupo j y g = vector de efectos de los grupos genéticos.
Se han considerado dos grupos genéticos diferentes de acuerdo con el origen genético de los animales. En un grupo se han incluido los antecesores desconocidos de los 26 animales hiperprolíficos de ambos sexos importados de Francia desde 1994 (HP), incluyendose en el otro (ST) los antecesores desconocidos del resto de los reproductores. La media de la contribución genética de ambos grupos a los animales nacidos desde 1993 a 1997 han sido obtenidos a partir de la matriz Q y se presentan en la Figura 1.
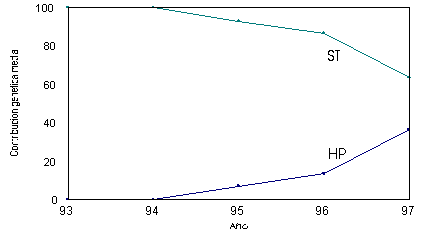
Figura 1. Media de la contribución genetica de los distintos grupos por año.
La proporción de genes del grupo HP aumenta desde el comienzo de la importación, año 1994, de 0 a 0.365 en el año 1997 en las cerdas analizadas.
En el muestreo de Gibbs se realizaron 765.000 iteraciones, descartándose las 5.000 iniciales, con un intervalo de muestreo de 1 / 76, salvándose 10.000 muestras de cada parámetro de interés. El número efectivo de muestras oscila entre 895 (c2) y 8.224 (efecto grupo: GHP – GST).
RESULTADOS Y DISCUSIÓN
Los resultados correspondientes a los principales efectos fijos confirman el importante efecto del ordinal de parto. Las medias (y d.t.) del efecto del ordinal de parto respecto al primero son: 1.55 (0.10), 2.08 (0.12), 1.84 (0.13), 1.55 (0.15) y 1.07 (0.15) lechones. Las medias (y d.t.) de las diferencias en tamaño de camada de los cruces por LD y PT respecto a los apareamientos LW en pureza son 0.30 (0.17) y 0.06 (0.18) respectivamente.
En la Tabla 2 se presentan los principales estadísticos de las distribuciones marginales posteriores de los componentes de varianza, heredabilidad y coeficiente del ambiente permanente, asi como de la diferencia entre grupos genéticos para el tamaño de camada (GHP – GST).
Tabla 2. Estimas de las distribuciones marginales posteriores de la varianza aditiva (s 2a), permante (s 2c), residual (s 2e),fenotípica (s 2p), heredabilidad (h2), coeficiente de ambiente permanente (c2) y de la diferencia entre grupos genéticos (GHP – GST).
Media |
Moda |
Mediana |
d.t. |
95% MDP |
|
s 2ª |
0.810 |
0.795 |
0.802 |
0.169 |
0.493 – 1.141 |
s 2c |
0.334 |
0.202 |
0.317 |
0.173 |
0.046 – 0.654 |
s 2e |
8.645 |
8.638 |
8.645 |
0.177 |
8.292 – 8.991 |
s 2p |
9.788 |
9.811 |
9.789 |
0.172 |
9.453 – 10.124 |
h2 |
0.083 |
0.080 |
0.082 |
0.017 |
0.051 – 0.116 |
c2 |
0.034 |
0.021 |
0.032 |
0.018 |
0.005 – 0.067 |
Efecto Grupo |
|||||
GHP - GST |
0.769 |
0.720 |
0.764 |
0.360 |
0.080 – 1.490 |
La mayoria de las distribuciones marginales de los parámetros estudiados fueron unimodales y prácticamente simétricos con respecto a la moda, siendo los valores posteriores de la media, mediana y moda muy similares y de acuerdo con lo aparecido en la literatura. Sin embargo, las correspondientes a los parámetros genéticos s 2c y c2 presentaron varios picos y un intervalo del 95 % de máxima densidad posterior muy amplio, lo que refleja el escaso número de cerdas (1578) con partos repetidos.
Por otro lado, se confirma la superioridad genética de los animales HP respecto a los animales ST lo que se pone de manifiesto al no incluir el intervalo del 95% MDP el valor nulo. La diferencia media estimada (0.77 lechones más en el grupo HP) es ligeramente inferior a la obtenida en Francia tanto en núcleos de selección como en granjas de multiplicación donde la diferencia era de un lechón nacido vivo (Herment y col., 1994). Ello hace aconsejable el empleo de esta línea aunque el balance definitivo requiere valorar su posible retraso genético para caracteres de composición corporal.
REFERENCIAS BIBLIOGRÁFICAS
HERMENT, A., RUNAVOT, J.P. y BIDANEL, J.P. 1994. Journées Rech. Porcine en France. 26: 315 – 320.
NOGUERA, J.L., ALFONSO, L., BABOT, D., PÉREZ-ENCISO, M. Y ESTANY J. Proc. 6th WCGALP. 23: 660 – 663.
WANG, C.S., RUTLEDGE, J.J. y GIANOLA, D. 1994. Genetics Selection Evolution. 26: 91 – 115.