BÚSQUEDA DE QTLS EN EL CROMOSOMA 6 PARA GRASA INTRAMUSCULAR Y PIGMENTOS EN UN CRUCE F2 IBÉRICO X LANDRACE
C. Óvilo 1, M. Pérez-Enciso 2, C.Barragán 1, A. Clop 3, M. Toro 1,M.C. Rodríguez 1, M. Oliver 4, C. Barboni 4, J.L. Noguera 2.
1 Departamento de Mejora Genética y Biotecnología (INIA, Madrid), 2 Area de Producció Animal (Udl-IRTA, Lleida), 3 Unitat de Genètica i Millora (UAB, Bellaterra), 4 Centre de Tecnología de la Carn (Monells, Girona)
INTRODUCCIÓN
La detección de marcadores moleculares asociados con calidad de la carne es particularmente interesante ya que su importancia es creciente en los programas de mejora. La utilización de marcadores moleculares para la detección de QTLs se ha visto enormemente facilitada gracias al formidable desarrollo de los mapas genéticos. En la actualidad el mapa genético porcino contiene aproximadamente 1800 marcadores de los que unos 250 son genes identificados.
En el presente trabajo presentamos resultados relativos al cromosoma 6 para dos caracteres: porcentaje de grasa intramuscular (GRIN) y cantidad de pigmentos hemo (PIGM) obtenidos en un cruce experimental F2 entre la línea Guadyerbas de cerdo Ibérico (IB) , y la Landrace (LD) de Nova Genètica (The IBMAP Consortium, 1998). En una comunicación paralela se presentan los resultados relativos al cromosoma 4 (Clop et al., 1999).
MATERIAL Y MÉTODOS
Material animal y caracteres. El pedigrí disponible consta de 3 machos IB, 31 hembras LD, seis machos y 73 hembras F1 y 577 animales F2. Aquí presentamos los resultados correspondientes a 200 (GRIN) y 204 (PIGM) animales F2, elegidos por su pertenencia a las familias de hermanos más grandes (28 para GRIN y 29 para PIGM). El manejo de los animales F1 y F2 fue en condiciones comerciales habituales para la línea LD. El sacrificio se llevó a cabo en 4 (GRIN) y 5 (PIGM) lotes a 175.1 + 0.3 días y un peso de canal promedio de 75.4 + 0.7.
La medida de GRIN se realizó mediante la técnica NIT (Gispert et al, 1997) y la del carácter PIGM según el método Hornsey (1956) modificado que expresa los m g de hematina ácida / g. de tejido fresco. Ambas medidas se tomaron en el músculo longissimus thoracis. En un estudio preliminar (Serra et al, 1998) para cuantificar las diferencias fenotípicas entre las dos poblaciones se encontró que para ambos caracteres éstas eran muy significativas.
Marcadores moleculares. Los animales parentales, F1 y F2 se genotiparon para 7 microsatélites del cromosoma 6 analizados en un secuenciador automático (ABI PRISM 310 Genetic Analyzer), utilizando cebadores fluorescentes amablemente distribuidos por el coordinador del U.S. Pig Genome.
Análisis estadístico. La construcción del mapa se realizó utilizando el programa Cri-Map (Green, et al., 1990).El contenido informativo (CI) fue calculado para cada marcador utilizando toda la información molecular disponible simultáneamente, siguiendo la metodología de Knott et al (1998) Asimismo se calculó también el índice de informatividad de Ron (IR; Ron et al, 1995). El modelo para el análisis de QTLs incluyó el sexo, lote y genotipo del QTL como efectos fijos, así como peso de canal como covariable. El análisis se realizó mediante mapeo de intervalos con regresión utilizando el método de Haley et al. (1994), que permite incluir la información de todos los marcadores cuando no son completamente informativos. Se asume que existe un QTL con alelos alternativos fijados en cada raza con valores a y -a para los homocigotos de cada raza, y d para los heterocigotos. El modelo que incluye el QTL se compara con el modelo sin QTL cada cM mediante un test F clásico.
RESULTADOS
Mapa de ligamiento. El mapa obtenido para el cromosoma 6 es parecido al obtenido por otros autores e indica una longitud de 156.9 cM para la hembra y de 129.1 cM para el macho. En la Tabla 1 se presentan los resultados relativos a la informatividad y posición de los microsatélites. Se observa que, con excepción de los microsatélites s0035 y s0228, la informatividad fue muy alta.
Tabla 1. Microsatélites utilizados: Posición en el mapa, número de alelos, índice de informatividad de Ron y contenido informativo.
Marcador |
|||||||
S0035 |
Sw1057 |
S0087 |
Sw316 |
S0228 |
Sw1881 |
Sw2419 |
|
Posición |
0 |
44 |
58 |
81 |
92 |
104 |
139 |
I.R. 1 |
0.79 |
0.85 |
1 |
0.85 |
0.60 |
0.75 |
0.92 |
Nº alelos |
4 |
4 |
6 |
7 |
6 |
8 |
8 |
C.I. 2 |
0.56 |
0.86 |
0.96 |
0.89 |
0.42 |
0.56 |
0.91 |
1.- I.R: Indice de Rón; 2.-C.I: Contenido Informativo
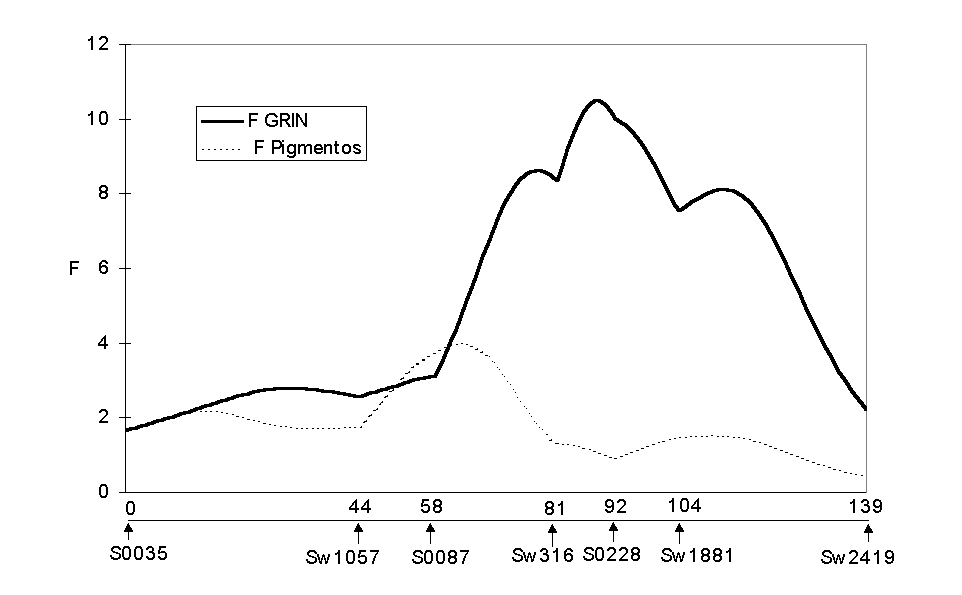
Análisis de QTLs. Los valores medios para ambos caracteres en la población F2 fueron 1.07+0.22 (GRIN) y 31.67+2.41 (PIGM). En la Figura 1 se presentan los valores del cociente F cada cM. Las estimas de a y d se calcularon para la mejor estima de la posición. Para GRIN se ha encontrado evidencia de un QTL en la posición 88, próxima al microsatélite s0228, con un máximo claramente significativo. El valor F (2 y 199 grados de libertad) es de 10.51, P<0.0001. Las estimas de los efectos aditivo y dominante fueron a = 0.24 + 0.06 y d =-0.24 + 0.10. Para PIGM se detectó un máximo en la posición 63, próxima al microsatélite s0087, F=3.99 (P<0.01). Los valores de a y d fueron 1.64 + 0.73 y -1.77+1.08, respectivamente. Tanto para GRIN como PIGM, el alelo Ibérico es el que incrementaría el valor del carácter.
Aunque los niveles nominales de significación son sustanciales para ambos caracteres, los umbrales de significación deben corregirse por el hecho de que todo el genoma va a ser considerado en última instancia. No se han realizado los cálculos pertinentes, pero trabajos de otros autores en experimentos de tamaño similar a éste sitúan el valor del umbral alrededor de 8, por lo que únicamente para GRIN la evidencia de un QTL sería concluyente. En el cromosoma 6 se conocen actualmente 47 genes, algunos de ellos muy interesantes, por ejemplo, el h-FABP que afecta al porcentaje de grasa intramuscular cuya posición no es conocida y otros genes candidatos para caracteres de engrasamiento como el gen del receptor de la leptina (LEPR) y el gen de la apolipoproteína E (APOE).
Figura 1. Perfil del valor F para distintas posiciones de un único QTL que afecte a grasa intramuscular (GRIN) y pigmentos (PIGM). El nivel de significación genómico es aproximadamente F = 8.
Agradecimientos
A Pere Borràs, Eva Ramells y en general a todo el personal de Nova Genètica y del matadero de Copaga por su inestimable cooperación. A Agustí Quintana y Meritxell Arqué por la ayuda en la toma de muestras. A Chris Haley por los programa de análisis. Al SIA Dehesón del Encinar que facilitó los parentales Guadyerbas. Trabajo financiado por la CICYT (proyecto AGF 96-2510).
REFERENCIAS BIBLIOGRAFICAS
Gispert et al (1997) Eurocarne 61, 27-32.
Green et al (1990) CriMap, Version 2.4. Washington University School of Medicine, St Haley et al (1994) Genetics 136, 1195-1207.
Knott et al (1998) Genetics 149, 1069-1080.
Ron et al (1995) Animal Genetics 26, 439-441
Serra X. et al (1998) Livest. Prod. Sci. 56: 215-223.
The IBMAP Consortium (1998) Proc. 6th World Congress on Genetics Applied to Livestock production 26, 316-319.