ANÁLISIS DE LA HETEROGENEIDAD ALÉLICA DEL GEN DE LA MIOSTATINA EN DIFERENTES RAZAS BOVINAS EUROPEAS
Miranda, ME.; Royo, L.; García-Atance, P.; Cañón, J.; Georges1, M.; Ménissier2, F.; Grobet1, L.; Bosher3, MY.; Dunner, S.
Laboratorio de Genética. Dpto Producción Animal. Facultad de Veterinaria. 28040 Madrid.1 Service de Genétique. Faculté de Médecine Vétérinaire. 20 Blvd Colonster. 4000 Liège. Bélgica.2 INRA SGQA. 78352 Jouy-en-Josas. Francia. 3 Labogena. 78352 Jouy-en-Josas. Francia
INTRODUCCIÓN
La hipertrofia muscular bovina es un fenómeno que aparece en diferentes razas europeas y cuya causa se debe a las mutaciones que afectan el gen de la miostatina descrito recientemente (McPherron y col., 1997; Grobet y col., 1997; Kambadur y col., 1997; Smith y col., 1997). Durante la embriogénesis y el posterior desarrollo, intervienen unos factores de crecimiento (Growth and Differentiation Factors (GDF)) dentro de los que se encuentra la miostatina. Esta y otras proteínas GDF pertenecen a la superfamilia de los TGFb (Transforming Growth Factors) por su parecido en su secuencia aminoacídica primaria y por codificar unos polipéptidos que se unen por puentes disulfuro. La miostatina está codificada por un gen compuesto por tres exones y cuya pérdida de función es la responsable de producir el desarrollo muscular exagerado al que se suele llamar hipertrofia muscular (Grobet y col., 1997). Los animales de la especie bovina con este fenotipo presentan un desarrollo muy superior de la masa muscular, una disminución significativa en la cantidad de grasa y suelen llamarse coloquialmente animales "culones, culard, etc". La pérdida de función se produce a consecuencia de unas mutaciones (sustituciones o delecciones) que alteran el marco de lectura, produciendo la aparición de un codon stop o bien son responsables de un cambio aminoacídico que produce la alteración de la proteína. Hasta ahora se han descrito seis mutaciones de estas características (Grobet y col., 1998; Cappuccio y col., 1998) además de otras seis mutaciones silenciosas que no causan alteraciones en la proteína final. La necesidad de estudiar qué mutaciones aparecen en qué razas para entender el distinto comportamiento de la hipertrofia muscular es el objeto de un proyecto coordinado que incluye diferentes razas francesas, españolas y belgas (Proyecto Europeo de Demonstración PL980421).
En el marco de este proyecto, es necesario poner a punto una técnica que permita combinar el diagnóstico de todas las mutaciones conocidas así como la detección de otras nuevas. Una de las técnicas de elección es la técnica PCR-SSCP (Polymerase Chain Reaction-Single Strand Conformation Polymorphism -Orita y col., 1989-) que se basa en la migración diferencial de fragmentos PCR desnaturalizados que adquieren conformaciones intracatenarias distintas en función de su composición nucleotídica. En este trabajo mostramos un ejemplo (ya que al ser datos preliminares, todavía no se ha conseguido en su totalidad) de la utilización de PCR-SSCP para combinar varios fragmentos en multiplex y resolver los patrones de migración que nos permitirán analizar todas las muestras e identificar nuevas mutaciones por secuenciación de los patrones anómalos.
MATERIAL Y MÉTODOS
Muestras de sangre. Las muestras de sangre corresponden a animales incluidos en el proyecto Euromh (Proyecto Europeo de Demonstración PL980421), pertenecientes a distintas razas, tanto españolas: Asturiana, Rubia Gallega y
Pirenaica, como francesas: Parthenaise, Maine-Anjou, Charolés, Limousine, INRA95, Gasconne, Bazadaise o belgas: Blanco Azul Belga y Blanc Bleu Belge mixte además de algunos animales de raza Piamontesa.
La técnica se puso a punto utilizando como referencia a algunos animales de mutaciones conocidas. La extracción de ADN se realizó mediante el método de Watt y Watt (1992).
Diseño de cebadores. Basándonos en la secuencia del gen (datos no publicados), diseñamos 4 pares de cebadores para amplificar el exón 2 del gen de la miostatina y sus secuencias intrónicas flanqueantes en 4 fragmentos solapantes entre sí. De esta manera quedaban incluidas las mutaciones y polimorfismos genéticos conocidos hasta el momento que se localizan en el intrón 1 y exón 2 obteniendo un total de 4 fragmentos : AB1, AB2, CD y EF. La optimización de las condiciones para los fragmentos CD y EF, que incluyen 2 y 1 mutaciones ya conocidas respectivamente, se llevó a cabo con la incorporación de nuevos controles positivos, obtenidos mediante el diseño de un nuevo cebador forward para cada uno de estos dos fragmentos idéntico al original salvo en un nucleótido, de modo que incorporamos a la secuencia una mutación de tipo transición (ofrece mayores dificultades de detección) de forma artificial.
Multiplex-PCR. Se realizó la amplificación simultánea de 2 fragmentos en una sola reaccion: AB1 y EF, en un volumen de 50 m l conteniendo 100ng de ADN genómico, 20 pmol de cada cebador del fragmento AB1 y 3.6 pmol de los correspondientes al EF(también el cebador artificialmente mutado), 2mM de Cl2Mg, 200 m M de dNTPs y 1 U de Taq Polimerasa. El programa de PCR constaba de una primera desnaturalización a 95° C durante 5 min y 30 ciclos de 95° C 1min, 55° C 1min y 72° C 1min con una última fase de extensión a 72° C 5 min.
Análisis SSCP. Los productos PCR se mezclan con un igual volumen (3m l) de tampón de carga (.05% de xylene-cyanole, .05% azul de bromofenol, 5.5 mM de EDTA, pH 8.0, en formamida). La mezcla se desnaturaliza a 95° C durante 5 minutos y se mantiene en hielo 2 min., antes de su carga en el gel en una cubeta de electroforesis PenguinTM Dual-Gel, Water-cooled Electrophoresis System (OWL Scientific Inc., Woburn, MA), con control de temperatura COOLINE Plus 8-30e. La carga de la PCR dúplex permite visualizar simultáneamente en el gel SSCP los fragmentos AB1 y EF, distinguiendo los diferentes haplotipos que se generan para cada animal.
Los parámetros que se alteraron para conseguir las mejores condiciones de detección fueron el ratio Acrilamida:N,N´-methylene-bis-acrilamida, la concentración del gel, la presencia/ausencia de glicerol y la temperatura de electroforesis.
RESULTADOS Y DISCUSIÓN
Las mejores condiciones halladas para la detección de mutantes tanto conocidos como desconocidos en estos dos fragmentos simultáneamente son 16% de concentración en el gel, ausencia de glicerol, TBE 0.5 X y 10° C de temperatura. En estas condiciones diferenciamos todos los haplotipos presentes en los dos fragmentos y previamente identificados en los animales utilizados como referencia, además de las muestras artificialmente mutadas
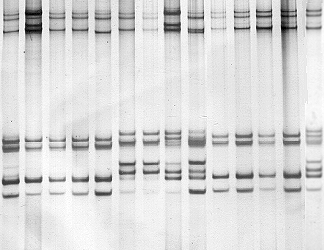
En la figura 1 se pueden ver diferentes individuos con sus haplotipos correspondientes reseñados en la tabla adjunta. Este tipo de análisis se puede realizar con todos los fragmentos simultáneamente, permitiendo la lectura del haplotipo completo de cada uno de los animales analizados.
REFERENCIAS BIBLIOGRAFICAS
Cappuccio y col. (1998). XXVI Int. Conf.on Animal Genetics. ISAG. Nueva Zelanda.
Grobet y col. (1997). Nature Genetics 17: 71-74.
Grobet y col. (1998). Mammalian Genome 9: 210-213.
Kambadur y col. (1997). Genome Research 7: 910-915.
Levin y col. (1993). Mutation Research, 288, 85-92.
McPherron y col. (1997). Nature 387: 83-90.
Orita y col. (1989). Genomics, 5, 874-879.
Smith y col., (1997). Mammalian Genome 8: 742-744.
Watt y Watt (1992). Molecular Ecology 1: 131-132.