ESTUDIO GENÉTICO mediante MARCADORES microsátelites
DE LAS POBLACIONES DE CABRA MONTÉS
N. Jiménez1, J. Folch2, A. Fernández-Arias2, J. Guiral3, A. Sánchez1
1 Unitat de Genètica i Millora. Facultat de Veterinària. Universitat Autònoma de Barcelona. 08193 Bellaterra (Barcelona).
2 Unidad de Tecnología en Producción Animal. Servicio de Investigación Agroalimentaria (Diputación General de Aragón). Apdo. 727. 50080 Zaragoza. 3 Servicio de Vida Silvestre. Edificio Pignatelli (Diputación General de Aragón). 50080 Zaragoza.
INTRODUCCIÓN
La cabra montés (Capra pyrenaica) es un ungulado localizado en España de la que se consideran 4 subespecies (Cabrera, 1911) : Capra pyrenaica victoriae, Capra pyrenaica hispanica, Capra pyrenaica lusitanica (extinguida en el siglo XIX) y Capra pyrenaica pyrenaica o bucardo (localizada en el Pirineo Central).
La existencia de escasos ejemplares localizados de bucardo, y su inclusión en el Catálogo Nacional de Especies en Vías de Extinción, impulsó a la Diputación General de Aragón a poner en marcha un plan de recuperación en 1993 (Guiral et al. 1997) que contempla, entre otras acciones, precisar las características genéticas de este animal en relación a las otras subespecies. La finalidad es determinar la población más polimórfica y parecida genéticamente para cruzarse con ella o, eventualmente, ser candidata para ser introducida en el ecosistema pireanaico.
Estudios anteriores, realizados en Francia y España (Manceau, 1997; Villalta et al., 1997), basados en ADN mitocondrial apoyaron la importancia de preservar la Capra pyrenaica pyrenaica al ser una subespecie monofilética y recomendaron la utilización de marcadores nucleares de ADN para completar el estudio genético del bucardo.
Por ello, en el presente estudio se han utilizado marcadores moleculares del tipo microsatélite, repeticiones cortas de ADN en tándem, distribuidas aleatoriamente por todo el genoma y con un elevado nivel de polimorfismo en la mayor parte de organismos eucariotas en los que han sido descritos.La batería de microsatélites utilizados se ha puesto a punto a partir de marcadores descritos en la especie caprina y conservados evolutivamente en las especies bovina y ovina. Esta conservación evolutiva en rumiantes los convierte en buenos candidatos para ser analizados en Capra pyrenaica pyrenaica.
MATERIAL Y MÉTODOS
Las muestras analizadas corresponden a las diferentes poblaciones de interés para la realización de este estudio, incluida una muestra de cabra doméstica que ha servido de control. El total de muestras ha sido de 59 repartidas de la siguiente manera:
Capra pyrenaica victoriae : 9 muestras de hígado de la población de Batuecas y 14 muestras de hígado de la población de Gredos.
Capra pyrenaica hispanica : 6 muestras de sangre de la población de Beceite, 12 muestras de hígado de la población de Tortosa, 5 muestras de hígado de la población de Muela de Cortés y 5 muestras de sangre de la población de Sierra Nevada.
Capra pyrenaica pyrenaica : 1 muestra de sangre y 1 de pelo de la población del Parque Nacional de Ordesa.
Capra ibex (se eligió por su posible proximidad genética con el bucardo): 5 muestras de sangre de la población de Vanoisse (Francia).
Capra hircus (control) : 1 muestra de sangre.
La extracción de ADN se realizó por métodos convencionales con extracción fenol-cloroformo.
El ADN obtenido fue cuantificado en gel de agarosa 0.8% y posteriormente con el procesador de imágenes Bioprint Version 97 y el programa Bio 1D (Vilber Lourmat Biotechnology).
Marcadores moleculares
Se realizó PCR de los ADNs para una batería de 13 microsatélites ( ILST 0005, ETH 152, ETH 10, Hel 9, ETH 225, CSSM 66, INRA 035, INRA 005, INRA 063, CHI AE 54, INRA 023, INRA 032 e INRA 037) con cebadores marcados con fluorescencia y optimizados para Capra pyrenaica y Capra ibex.
Condiciones de PCR : 1.5mM de MgCl2 para todos los microsatélites excepto INRA 023 e INRA 023 (2mM) e CHI AE 54 (2.5mM), 250 m M de cada dNTP para todos los microsatélites excepto CHI AE 54 (100m M), 0.05 m M de cada cebador, 100-200 ng de ADN y 0.5 unidades de Taq ADN polimerasa (Gibco BRL) (excepto CHI AE 54 que eran 0.125 unidades) en un volumen final de 20 m l excepto CHI AE 54 donde el volumen final fue de 50 m l. Las condiciones de amplificación fueron: desnaturalización inicial de 5’ a 94ºC seguida de 29 ciclos de 20’’ a 94ºC (excepto CHI AE 54 con 27 ciclos de 1' a 94ºC), hibridación de 20’’ a 55ºC (excepto INRA 023 e INRA 032 que eran 20'' a 50ºC y CHI AE 54 2' a 50ºC), extensión de 30’’ a 72ºC (excepto CHI AE 54 que era 1' a 72ºC sin extensión final), el resto de microsatélites tenían una extensión final de 5’ a 72ºC.
El análisis de los fragmentos amplificados se realizó mediante un equipo automatizado de electroforesis capilar (Genescan ABI Prism 310 de Perkin Elmer) para establecer su tamaño y el genotipo de las muestras.
El cálculo de las distancias genéticas, la estima de la variabilidad genética y la construcción de dendogramas se realizó mediante el programa informático Biosys-1 versión 1.7 (Swofford & Selander, 1989).
RESULTADOS Y DISCUSIÓN
Los resultados obtenidos con los 13 marcadores analizados muestran:
a) La ausencia total de polimorfismo para el panel de microsatélites en la muestra de bucardo analizada. Los 13 marcadores estudiados son monomórficos en el ejemplar analizado. Este resultado es especialmente relevante si tenemos en cuenta el elevado nivel de polimorfismo de los marcadores analizados (la heterozigosidad, H, en la muestra de cabra doméstica analizada era de 0.46). Resultados preliminares del análisis de los pelos del segundo ejemplar de bucardo (presumiblemente el último vivo de la subespecie) apuntan a una situación equivalente. De este segundo ejemplar sólo se pudieron analizar 8 marcadores, monomórficos todos ellos, pero con un alelo distinto al encontrado en el otro ejemplar analizado para el marcador INRA 032.
Los bajos valores de variabilidad (la H oscila entre 0.113 y 0.274) obtenidos en general reflejan probablemente la elevada consanguinidad y cuellos de botella que han sufrido estas poblaciones.
b) Tres de los 13 microsatélites analizados no han resultado informativos al ser monomórficos y presentar para todas las poblaciones de Capra pyrenaica y Capra ibex el mismo alelo fijado. Por este motivo se han reanalizado los resultados eliminando estos marcadores con un lógico aumento de la H.
Tabla 1 : Variabilidad genética de los 10 loci en todas las especies y subespecies
Especie-subespecie |
Tamaño medio de muestra por locus |
Nº medio de alelos por locus |
Porcentaje de loci polimórficos |
Heterozigosidad media |
|
Observ. |
Esperada HdyWbg |
||||
| C.p.victoriae | 23.00 |
2.3 |
80.0 |
0.239 |
0.308 |
| C.p.hispanica | 27.00 |
3.3 |
90.0 |
0.274 |
0.488 |
| C.p.pyrenaica | 1.0 |
1.0 |
0.0 |
0.000 |
- |
| C.ibex | 5.0 |
1.7 |
40.0 |
0.160 |
0.178 |
| C.hircus | 1.0 |
1.6 |
60.0 |
0.600 |
- |
c) Las distancias genéticas (D de Nei) obtenidas entre poblaciones oscilan entre 0.044 para las 2 poblaciones de Capra pyrenaica victoriae (Batuecas y Gredos) y 1.117 entre la población de Capra Ibex y el ejemplar de bucardo analizado.
d) El nivel de diferenciación observado entre el íbice alpino y el resto de poblaciones ibéricas es muy similar: 1.081 con Capra pyrenaica victoriae; 0.921 con Capra pyrenaica hispanica y 1.117 con el ejemplar de bucardo.
e) La distancia genética promedio entre el bucardo y las demás subespecies de Capra pyrenaica es de 0.493 con victoriae y de 0.532 con hispanica. Estos valores no resultan significativamente distintos de los observados entre Capra pyrenaica victoriae y Capra pyrenaica hispanica (0.316) y son de una magnitud equivalente o incluso inferior a la obtenida entre algunas poblaciones de la misma subespecie (0.543 es la distancia promedio entre las 4 poblaciones de Capra pyrenaica hispanica analizadas).
f) Los árboles filogenéticos obtenidos al agrupar los datos por subespecies confirman el origen monofilético del bucardo.
Figura 1 : Árbol filogenético para los 10 microsatélites informativos
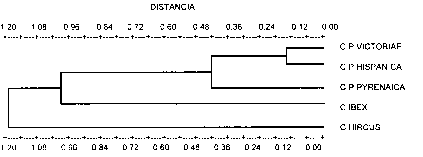
g) El análisis filogenético por poblaciones colocan al bucardo más cercano a las poblaciones de Capra pyrenaica hispanica, en especial las de Tortosa y Muela de Cortés, que serían las mejores candidatas para una hibridación con el ejemplar vivo de bucardo.
AGRADECIMIENTOS
El trabajo se ha realizado en el marco de un acuerdo de colaboración entre la Diputación General de Aragón y la Universitat Autònoma de Barcelona, con financiación a través del Proyecto Life: Programa de conservación de vertebrados amenazados del Pirineo.
REFERENCIAS BIBLIOGRÁFICAS
Amills, M. et al. 1996. Animal Genetics, 27: 435-436.
Blouin, M.S. et al. 1996. Molecular Ecology, 5: 393-401.
Cabrera A. 1911. The subspecies of Spanish Ibex. Prc. Zool. Soc. (Londres; Green).
Guiral, J. et al. 1997. The bucardo Capra pyrenaica pyrenaica recovery plan. 2nd World Conference on Mountain Ungulates. Aosta (Italia).
Manceau, V. 1997. Thèse Doc. Université Joseph Fourier – Grenoble I.
Moazami, K. et al. 1997. Animal Genetics, 28: 338-345.
Swofford, D. et al. 1989. BIOSYS-1 A computer program for the analysis of allele variation in population genetics and biochemical systematics.
Villalta, M. et al. 1997. Theriogenology, 47 (1): 410.